Las moras, dentro del género Rubus, son un fruto ampliamente apreciado por su sabor característico y alto contenido en antioxidantes y compuestos beneficiosos como las antocianinas. Su relevancia comercial ha crecido considerablemente, siendo uno de los cultivos frutales más importantes en diversas regiones del mundo, especialmente en los Estados Unidos. Sin embargo, la complejidad genética y poliploide de muchas variedades, particularmente las tetraploides, ha representado un desafío considerable para los científicos y mejoradores interesados en optimizar sus características agronómicas y fitosanitarias. El reciente desarrollo del primer ensamblaje genómico completo a nivel cromosómico y con resolución haplotípica para una selección de mora tetraploide primocane-fructífera y sin espinas, denominada BL1, marca un antes y un después en el estudio genético de esta especie. Esta herramienta supone un avance significativo frente a las limitaciones históricas derivadas de la naturaleza poliploide y compleja de estas moras, facilitando el análisis detallado de alelos, la identificación de genes clave y la comprensión de mecanismos moleculares asociados con importantes rasgos de interés agrícola.
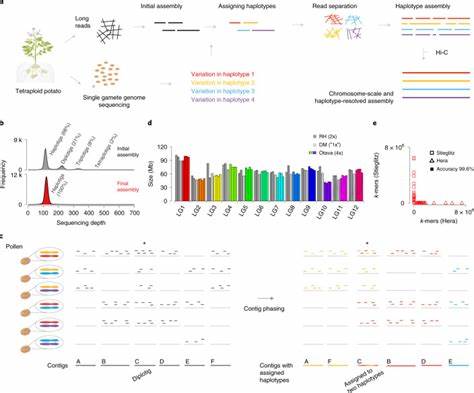
El ensamblaje se logró mediante la combinación de tecnologías avanzadas de secuenciación como Oxford Nanopore y el uso de scafolding con Hi-C, lo que permitió ordenar y asignar el 95% de las secuencias a 27 pseudo-cromosomas, abarcando una longitud aproximada de 919 megabases. La calidad y completitud del ensamblaje se confirmaron mediante indicadores como un elevado N50 y la recuperación de más del 98% de genes conservados, lo cual denota un gran nivel de detalle y precisión en la reconstrucción genómica. Uno de los aspectos destacables en la construcción de este genoma fue la resolución haplotípica, es decir, la capacidad de distinguir y ensamblar las cuatro copias homólogas propias del genoma tetraploide. Esta distinción es fundamental para conservar la información sobre variabilidad alélica y diferencias estructurales entre los cromosomas, información que se pierde en ensamblajes tradicionales que agrupan secuencias homólogas en consenso. De esta forma, la resolución haplotípica posibilita analizar con mayor profundidad la genética de la mora, lo que es crucial para entender la herencia de características como la ausencia de espinas o la fructificación en primocanes.
El genoma BL1 reveló que más del 58% de su contenido se compone de secuencias repetitivas, siendo las retrotransposones de tipo LTR las más abundantes. Además, se predijeron cerca de 88,000 genes codificantes de proteínas, de los cuales el 82% contaba con anotaciones funcionales que permiten inferir sus posibles roles biológicos. Este nivel de detalle asegura un recurso valioso para la identificación de genes implicados en características de alto interés hortícola. Entre las áreas de mayor interés identificadas a partir del genoma destaca la búsqueda de genes asociados con la ausencia de espinas, un rasgo indispensable para facilitar la cosecha, el manejo y la comercialización del cultivo. El locus responsable de la espinosidad se localizó en un tramo específico del cromosoma A4, donde se identificaron diversos genes relacionados con el desarrollo anatómico, la respuesta a estímulos químicos y el estrés.
En particular, se encontraron numerosos genes con dominios típicos de resistencias y de factores de transcripción conocidos por su papel en el desarrollo de tricomas, estructura de la cual derivan las espinas. Este hallazgo abre la puerta para la utilización de estos genes como marcadores moleculares en programas de mejoramiento orientados a obtener variedades sin espinas sin sacrificar otras cualidades agronómicas. La investigación también abarcó el estudio de rasgos revolucionarios como la fructificación en primocanes, que permite la producción de fruto en el mismo año de crecimiento del tallo, contrariamente a la fructificación en floricanas, lo que extiende la temporada de cosecha y mejora los rendimientos. Este rasgo fue sitiado en un segmento concreto del cromosoma A2, donde se detectaron más de 2,000 genes, varios de ellos relacionados con la regulación de la floración y vías de señalización hormonal. Entender estos mecanismos facilita diseñar estrategias para incorporar esta característica en nuevas variedades.
En cuanto a la resistencia y susceptibilidad a enfermedades, el análisis genómico permitió identificar un amplio repertorio de genes NLR (genes de resistencia basados en dominios nucleótido y leucina rica en repeticiones), cruciales para la defensa contra patógenos. Estos genes están distribuidos en varios cromosomas, con concentraciones mayores en regiones específicas que podrían ser foco de selección para mejorar la resistencia genética. La identificación de genes MLO, involucrados en la susceptibilidad a mildiu polvoriento, también brinda posibilidades para desarrollar variedades resistentes mediante técnicas como la edición génica. La expresión de estos genes fue analizada en diferentes tejidos y estados de desarrollo, proporcionando información para futuras intervenciones dirigidas. Otro aspecto biológico fundamental es la biosíntesis de antocianinas, pigmentos responsables del característico color oscuro y las propiedades antioxidantes de la mora.
El genoma BL1 facilitó la caracterización detallada de las rutas metabólicas y factores de transcripción que regulan la producción de estos compuestos durante las distintas etapas de maduración del fruto. Se observó cómo ciertos genes estructurales y reguladores activadores elevan su expresión a medida que el fruto madura y adquiere color, mientras que genes represores disminuyen. Este equilibrio es fundamental para controlar la calidad y características del fruto, lo que puede ser aprovechado para mejorar los perfiles de sabor, color y valor nutricional. La comparación genómica con otras especies de la familia Rosaceae evidenció la relación filogenética entre la mora tetraploide y otras especies como fresas, frambuesas y rosas. Esta información contribuye a comprender la evolución genómica y la diversificación de rasgos de interés comercial.
Además, estudiar las variaciones en los genomas de diferentes cultivares y selecciones permitió identificar millones de variantes genéticas, entre SNPs e indels, algunas de las cuales están asociadas a características tales como resistencia a enfermedades, hábitos de fructificación y presencia o ausencia de espinas. Este nivel de detalle en la caracterización genética representa una base sólida para la aplicación de técnicas avanzadas de mejoramiento, incluyendo la selección asistida por marcadores y la edición genética. Gracias al ensamblaje de alta calidad y a la resolución haplotípica, ahora es posible diseñar estrategias más precisas para combinar rasgos deseables, acelerar la obtención de nuevos cultivares y responder con mayor eficacia a desafíos agronómicos y de mercado. En cuanto a la metodología empleada, la combinación entre la longitud y profundidad de secuenciación mediante tecnologías nanopore, la protección de las interacciones cromosómicas con Hi-C, y el uso de métodos bioinformáticos avanzados para ensamblaje y anotación, fueron decisivos para lograr este nivel de calidad en el genoma. La integración de datos transcriptómicos de diversas fuentes permitió validar y mejorar la predicción génica, así como estudiar patrones de expresión en tejidos clave y etapas de desarrollo.
Además de la relevancia para la investigación y la mejora genética, este genoma es un recurso invaluable para la comunidad hortícola, ya que facilita la identificación de candidatos génicos relacionados con rasgos de producción, calidad y resistencia a enfermedades. Tess nuevos conocimientos abren nuevas vías para el análisis molecular, la biotecnología y la agricultura sostenible aplicada a las moras. La posibilidad de desarrollar moras sin espinas, con un ciclo de fructificación más corto y mejor resistencia a enfermedades, puede revolucionar la producción comercial, reducir costos y mejorar la satisfacción del consumidor. La disponibilidad pública del archivo genómico BL1, junto con los datos complementarios de cultivares y selecciones representativas, constituye una plataforma esencial para investigadores y mejoradores. Permite no solo explorar la diversidad genética sino también implementar herramientas moleculares para la aceleración de programas de selección y la incorporación de nuevos caracteres.
Asimismo, favorece la transferencia de conocimientos a cultivos relacionados dentro de la familia Rosaceae. En conclusión, el ensamblaje cromosómico y la resolución haplotípica del genoma de la mora tetraploide BL1 representan un avance profundo en la comprensión genética de esta especie compleja. Estas investigaciones promueven el desarrollo de variedades mejoradas que combinan calidad de fruto, facilidad de cultivo y resistencia a enfermedades, prioritarios para la expansión comercial y la sostenibilidad productiva. La integración de estos recursos genómicos con enfoques de biología molecular, genómica funcional y mejoramiento dirigirá un futuro prometedor para el cultivo de mora en todo el mundo.