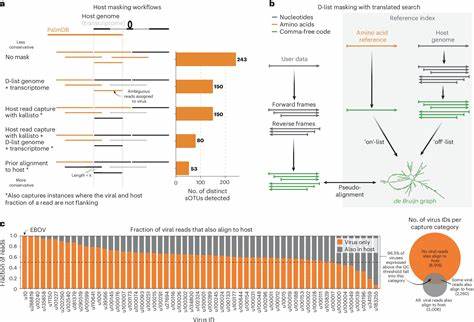
En el ámbito de la biología molecular y la bioinformática, la identificación precisa y eficiente de virus en muestras biológicas ha sido un desafío constante, especialmente cuando se trata de virus de ARN, que son responsables de numerosas enfermedades infecciosas. Recientemente, un equipo de investigadores del Instituto Tecnológico de California (Caltech) logró desarrollar un software amigable para el usuario que permite detectar virus en datos de secuenciación de ARN con una precisión y facilidad sin precedentes. Esta innovación no solo abre nuevas puertas para el estudio profundo del mundo viral, sino que también tiene el potencial de revolucionar la forma en que se monitorean enfermedades emergentes y se investiga la influencia de virus en distintos procesos biológicos. La secuenciación de ARN es una herramienta poderosa que permite a los científicos analizar las moléculas de ARN presentes en una muestra. Tradicionalmente, cuando se secuencia ARN de tejido humano o animal, la mayor parte del material identificado es del propio organismo hospedante, mientras que la presencia de virus suele pasar desapercibida o incluso descartarse, siendo considerada como ruido o dato irrelevante.
Sin embargo, el software desarrollado en Caltech, basado en una evolución de la reconocida herramienta kallisto, busca precisamente aprovechar esta información aparentemente secundaria para detectar con precisión la presencia de virus, incluso aquellos que son inesperados o nuevos para la ciencia. Uno de los avances clave para lograr esta detección es el enfoque del nuevo algoritmo en una característica común y fundamental de los virus de ARN: la enzima ARN polimerasa dependiente de ARN (RdRp, por sus siglas en inglés). Esta enzima es esencial para que los virus de ARN se reproduzcan, ya que les permite copiar su material genético. Detectar la secuencia genética que codifica para esta enzima facilita la identificación de una gran variedad de virus, superando la barrera de la diversidad genética viral. Así, la herramienta puede reconocer más de 100,000 especies virales con un coste computacional mínimo, lo que la hace accesible para laboratorios con distintos niveles de recursos.
La facilidad de uso es un aspecto fundamental que distingue a este software de otras herramientas existentes. Diseñado pensando en los biólogos y no exclusivamente en especialistas en informática, este programa permite que cualquier investigador con datos de secuenciación de ARN pueda ejecutarlo sin necesidad de conocimientos técnicos avanzados en programación o bioinformática. Esto democratiza el acceso a la tecnología de detección viral y fomenta una mayor participación en la vigilancia y estudio del mundo viral. Además, la herramienta no solo identifica viralidad general, sino que puede analizar datos de secuenciación a resolución de células individuales, lo que constituye una innovación notable. Esta capacidad permite a los científicos observar qué células están infectadas y cómo el virus afecta la expresión génica en cada tipo celular, información crucial para entender enfermedades virales complejas y sus mecanismos.
Además de su utilidad en la investigación básica, este software tiene aplicaciones prometedoras en la vigilancia epidemiológica y la salud pública. La detección temprana y precisa de virus en diferentes muestras biológicas contribuye a la identificación rápida de brotes y el seguimiento de enfermedades emergentes o reemergentes. Durante pandemias o en el monitoreo de virus zoonóticos, disponer de una herramienta que acelere el análisis puede marcar la diferencia en la toma de decisiones sanitarias. Un aspecto importante que destaca del software es su capacidad para conservar lecturas genéticas ambiguas entre virus y hospedante, que normalmente serían descartadas en otros procesos de análisis. Estos datos, aunque complejos, son valiosos para estudios detallados y permiten un análisis más profundo a nivel molecular, posibilitando descubrir nuevas relaciones entre virus y sus hospedantes o incluso identificar virus previamente desconocidos.
El desarrollo de esta herramienta ha sido posible gracias a la integración de bases de datos especializados, como PalmDB, que aglutinan información viral extensa y actualizada. La combinación de datos robustos con algoritmos innovadores resulta en una plataforma potente, pero al mismo tiempo accesible, ideal para acelerar avances en virología, medicina, biología molecular y campos relacionados. En un contexto donde la cantidad de datos obtenidos mediante técnicas de transcriptómica y secuenciación masiva crece exponencialmente, contar con herramientas eficientes y user-friendly para interpretar esta información es fundamental. El volumen de datos puede llegar a ser abrumador si no se cuenta con soluciones que simplifiquen su manejo y permitan extraer conclusiones relevantes sin requerir un equipo de bioinformáticos altamente especializado. Por último, la implicación de esta tecnología en la investigación de enfermedades neurodegenerativas y otras patologías crónicas abre una nueva línea de interrogantes y posibles descubrimientos.